TCGA数据库
1 TCGA Code Table1.1 Data LevelsLevel NumberDefinition1Raw data2Normalized data3Aggregated data4Regions of Interest data0No Level1.2 Portion / Analyte CodesCodeDe...
1 TCGA Code Table
1.1 Data Levels
| Level Number | Definition |
|---|---|
| 1 | Raw data |
| 2 | Normalized data |
| 3 | Aggregated data |
| 4 | Regions of Interest data |
| 0 | No Level |
1.2 Portion / Analyte Codes
| Code | Definition |
|---|---|
| D | DNA |
| G | Whole Genome Amplification (WGA) produced using GenomePlex (Rubicon) DNA |
| H | mirVana RNA (Allprep DNA) produced by hybrid protocol |
| R | RNA |
| T | Total RNA |
| W | Whole Genome Amplification (WGA) produced using Repli-G (Qiagen) DNA |
| X | Whole Genome Amplification (WGA) produced using Repli-G X (Qiagen) DNA (2nd Reaction) |
1.3 Sample Type Codes
| Code | Definition |
|---|---|
| 01 | Primary Solid Tumor(原发性实体肿瘤) |
| 02 | Recurrent Solid Tumor(复发性实体肿瘤) |
| 03 | Primary Blood Derived Cancer - Peripheral Blood(原发性血源性癌症 - 外周血) |
| 04 | Recurrent Blood Derived Cancer - Bone Marrow(复发性血源性癌症 - 骨髓) |
| 05 | Additional - New Primary |
| 06 | Metastatic(转移肿瘤) |
| 07 | Additional Metastatic |
| 08 | Human Tumor Original Cells(肿瘤原始细胞) |
| 09 | Primary Blood Derived Cancer - Bone Marrow(原发性血源性癌 - 骨髓) |
| 10 | Blood Derived Normal |
| 11 | Solid Tissue Normal(癌旁组织) |
1.4 TCGA Study Abbreviations
| Study Abbreviation | Study Name |
|---|---|
| LAML | Acute Myeloid Leukemia |
| ACC | Adrenocortical carcinoma |
| BLCA | Bladder Urothelial Carcinoma |
| LGG | Brain Lower Grade Glioma |
| BRCA | Breast invasive carcinoma |
| CESC | Cervical squamous cell carcinoma and endocervical adenocarcinoma |
| CHOL | Cholangiocarcinoma |
| LCML | Chronic Myelogenous Leukemia |
| COAD | Colon adenocarcinoma |
| CNTL | Controls |
| ESCA | Esophageal carcinoma |
| FPPP | FFPE Pilot Phase II |
| GBM | Glioblastoma multiforme |
| HNSC | Head and Neck squamous cell carcinoma |
| KICH | Kidney Chromophobe |
| KIRC | Kidney renal clear cell carcinoma |
| KIRP | Kidney renal papillary cell carcinoma |
| LIHC | Liver hepatocellular carcinoma |
| LUAD | Lung adenocarcinoma |
| LUSC | Lung squamous cell carcinoma |
| DLBC | Lymphoid Neoplasm Diffuse Large B-cell Lymphoma |
| MESO | Mesothelioma |
| MISC | Miscellaneous |
| OV | Ovarian serous cystadenocarcinoma |
| PAAD | Pancreatic adenocarcinoma |
| PCPG | Pheochromocytoma and Paraganglioma |
| PRAD | Prostate adenocarcinoma |
| READ | Rectum adenocarcinoma |
| SARC | Sarcoma |
| SKCM | Skin Cutaneous Melanoma |
| STAD | Stomach adenocarcinoma |
| TGCT | Testicular Germ Cell Tumors |
| THYM | Thymoma |
| THCA | Thyroid carcinoma |
| UCS | Uterine Carcinosarcoma |
| UCEC | Uterine Corpus Endometrial Carcinoma |
| UVM | Uveal Melanoma |
- 01A:癌症组织
- 01B:福尔马林浸泡样品
- 02A:复发组织
- 06A:转移组织
一般只留下01A样品的肿瘤组织样品做差异表达和生存分析,并且对于重复样品,随机选取一个。
参考文章:
2 表达信息VS临床信息
- cases表示患者个数
- files表示样品个数
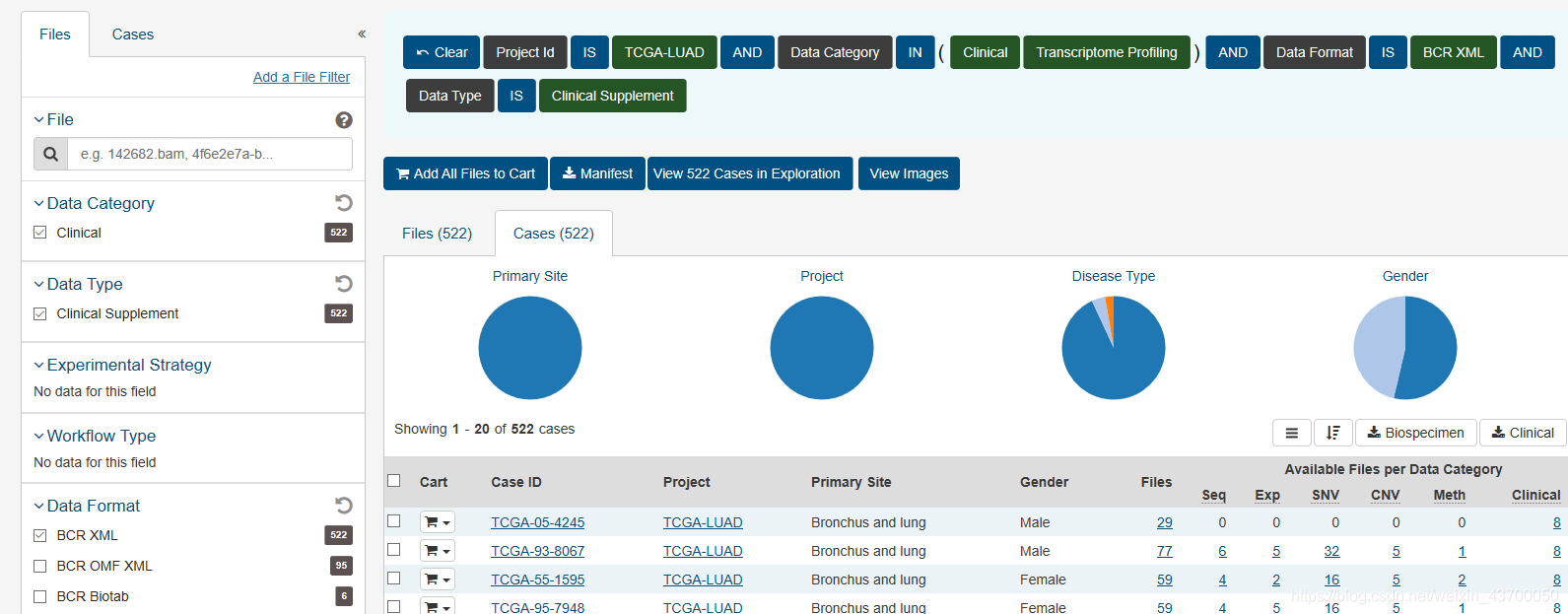
TCGA数据库不是所有患者都有表达信息,有的患者只有临床信息而没有表达信息。如TCGA-LUAD数据集,RNA-seq-data中有515个cases,594个files。而clinical-data却有522个cases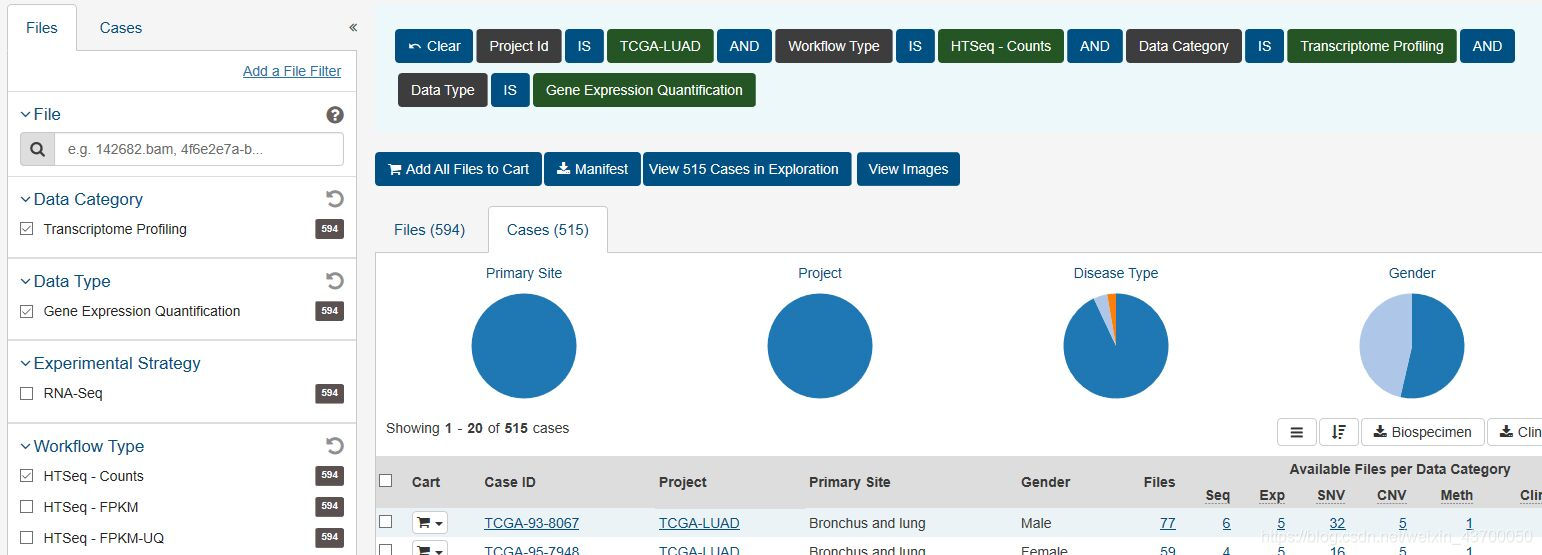
3 TCGA样本命名详解
在TCGA中,一个患者可能会对应多个样本,如TCGA-A6-6650可以得到3个样本数据:
- TCGA-A6-6650-01A-11R-1774-07
- TCGA-A6-6650-01A-11R-A278-07
- TCGA-A6-6650-01B-02R-A277-07
一般在做TCGA数据分析的时候样本名实际上只保留到前四个元素(以”-“分割),例如TCGA-A6-6650-01。所以实际上上示3个样本一般只保留一个。
| TCGA-A6-6650-01A-11R-1774-07 | ||
|---|---|---|
| TCGA | Project | 所有TCGA样本名均以这个开头 |
| A6 | Tissue source site | 组织来源编码,如A6就表示来源于Christiana Healthcare中心的结肠癌组织 |
| 6650 | Participant | 参与者编号 |
| 01 | Sample | 编号01~09为癌症组织,10~19表示正常对照 |
| A | Vial | 在一系列患者组织中的顺序,绝大多数样本该位置编码都是A; 很少数的是B,表示福尔马林固定石蜡包埋组织,已被证明用于测序分析的效果不佳,所以不建议使用-01B的样本数据 |
| 11 | Portion | 同属于一个患者组织的不同部分的顺序编号,同一组织会分割为100-120mg的部分,分别使用 |
| R | Analyte | 分析的分子类型,对应RNA |
| 1774 | Plate | 在一系列96孔板中的顺序,值大表示制板越晚 |
| 07 | Center | 测序或鉴定中心编码 |
所以现在看这三个样本:
TCGA-A6-6650-01A-11R-1774-07
TCGA-A6-6650-01A-11R-A278-07
TCGA-A6-6650-01B-02R-A277-07
其区别就在于,前两个使用的是患者的冰冻组织做的测序,而第三个用的是福尔马林固定石蜡包埋组织;而前两个样本的区别在于同一组织后续使用了不同的96孔板。
理解了命名规则及三者命名上的主要区别后,现在可以重点解决如何从一个患者的多个样本中挑选样本的问题了,首先排除TCGA-A6-6650-01B-02R-A277-07,因为是-01B,福尔马林固定石蜡包埋组织!剩下的两个:
- TCGA-A6-6650-01A-11R-1774-07
- TCGA-A6-6650-01A-11R-A278-07
先看看GDAC firehose遇到这种情况怎么解决,总结起来就是:
- 对RNA数据来说,Analyte为R的优先级最该,其次是R和T,而对于DNA层面的分析来说,D的优先级最高。
- 如果Analyte相同,那就选择Portion和/或Plate值更大的。 所以按照GDAC firehose的方法,最终保留TCGA-A6-6650-01A-11R-A278-07,因为其相对于TCGA-A6-6650-01A-11R-1774-07的板号(Plate)更晚。
然后是cBioPortal中的处理方式:
- 随机选择了一个,理由很简单啊,来源于同一个患者的癌组织样本差别不大。
参考文章:
本博客内容将同步更新到个人微信公众号:生信玩家。欢迎大家关注~~~
更多推荐
 已为社区贡献1条内容
已为社区贡献1条内容









所有评论(0)