学术速运|AlphaPeptDeep:一个预测蛋白质组学肽特性的模块化深度学习框架
题目:AlphaPeptDeep: a modular deep learning framework to predict peptide properties for proteomics文献来源:Nature Communications | (2022) 13:7238 1代码:https://github.com/MannLabs/alphapeptdeep简介:机器学习,特别是深度学习

题目:AlphaPeptDeep: a modular deep learning framework to predict peptide properties for proteomics
文献来源:Nature Communications | (2022) 13:7238 1
代码:https://github.com/MannLabs/alphapeptdeep
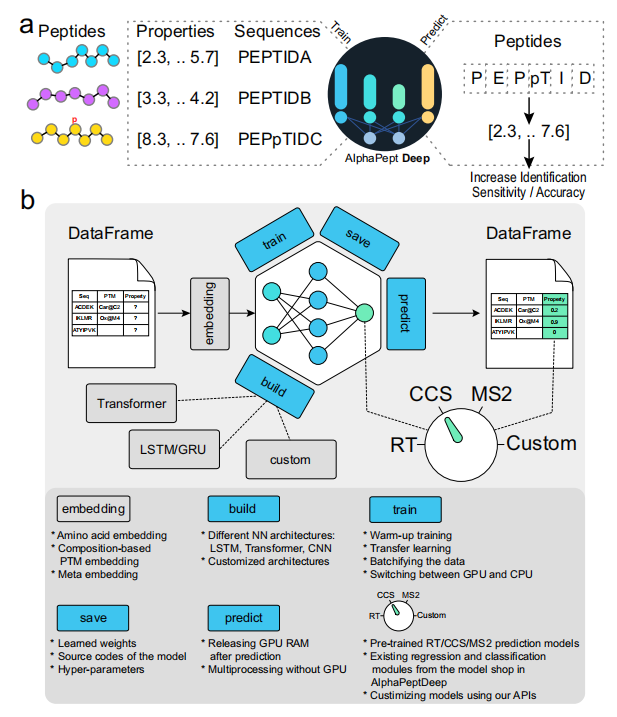
简介:机器学习,特别是深度学习(DL)在基于质谱(MS)的蛋白质组学中越来越重要。最近的DL模型可以从氨基酸序列中准确地预测肽的保留时间、离子迁移率和片段强度。然而,DL是一个发展非常迅速的领域,经常出现新的神经网络结构,这对蛋白质组学研究人员来说具有挑战性。在这里,我们介绍了AlphaPeptDeep,一个基于PyTorch DL库的模块化Python框架,它可以学习和预测肽的性质。它的特点是一个好操作,允许非专业人员在几行代码中创建模型。,即使只已知其化学成分,AlphaPeptDee也可以进行一般的翻译后修饰。迁移学习的广泛使用可以使用大数据集来满足特定实验条件下的模型的数据需求。用于预测保留时间、碰撞截面和碎片强度的AlphaPeptDeep模型至少与现有的工具相当。其他基于序列的特性也可以通AlphaPeptDeep进行预测,如进行HLA肽的识别。
主要内容:
更多推荐
 已为社区贡献6条内容
已为社区贡献6条内容









所有评论(0)