Nature Microbiology|Libusha Kelly组通过大语言模型改善病毒蛋白注释
大语言模型改善病毒蛋白注释Large language models improve annotation of prokaryotic viral proteinsResearch Article,2024-1-29,Nature Microbiology, [IF 28.3]原文链接:https://www.nature.com/articles/s41564-023-01584-8第一作者.
大语言模型改善病毒蛋白注释
Large language models improve annotation of prokaryotic viral proteins

Research Article,2024-1-29,Nature Microbiology, [IF 28.3]
原文链接:https://www.nature.com/articles/s41564-023-01584-8
第一作者:Zachary N. Flamholz
通讯作者:Libusha Kelly
通讯单位:美国阿尔伯特·爱因斯坦医学院系统与计算生物学系
- 摘要 -
宏基因组样本中的病毒基因组注释很差,这成为理解病毒多样性和功能的障碍。目前的注释方法依赖于基于比对的序列同源性方法,该方法受到特征病毒蛋白的缺乏和病毒序列之间的差异的限制。在这里,我们证明蛋白质语言模型(PLMs)可以捕获原核病毒蛋白质功能,从而使病毒序列空间的新部分能够被分配具有生物学意义的标签。当应用于全球海洋病毒组数据时,我们的分类器将病毒蛋白家族的注释部分扩展了 29%。在以前未注释的序列中,我们强调了定义海洋超微蓝藻中的移动元件的整合酶和锚定全球广泛传播的病毒元件的衣壳蛋白的鉴定。此外,改进的高级功能注释提供了一种表征不同病毒序列之间基因组组织相似性的方法。因此,蛋白质语言模型增强了病毒蛋白质的远程同源性检测,并可以作为现有方法的有益补充。
- 结果 -
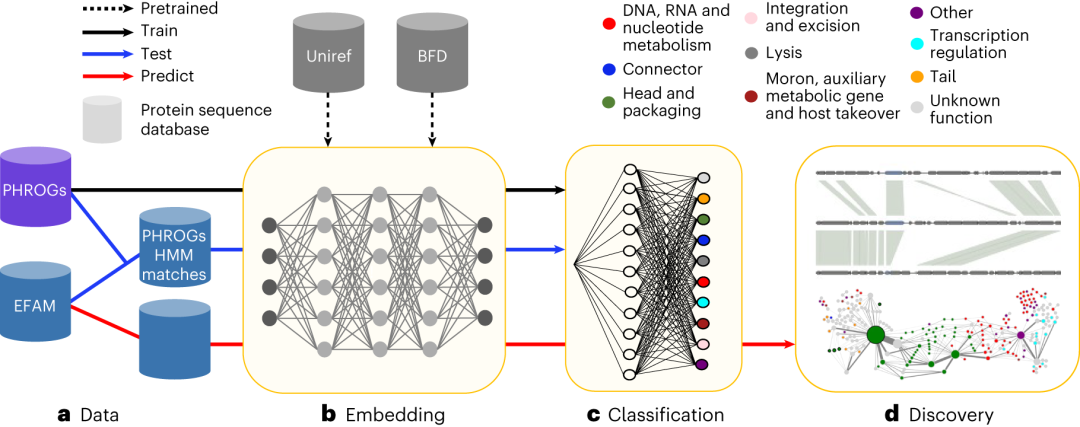
图1:使用 PLM 进行 VPF(病毒蛋白家族) 功能预测揭示了新的生物学特征。
a,VPF 是从手动策划的数据库 PHROGs 和 EFAM 中收集的。
b,使用预训练的 PLM 嵌入蛋白质序列。
c,嵌入被用于多级分类器的输入以进行高级函数预测。
d,未注释的 VPF 的分类器预测促使生物学发现。
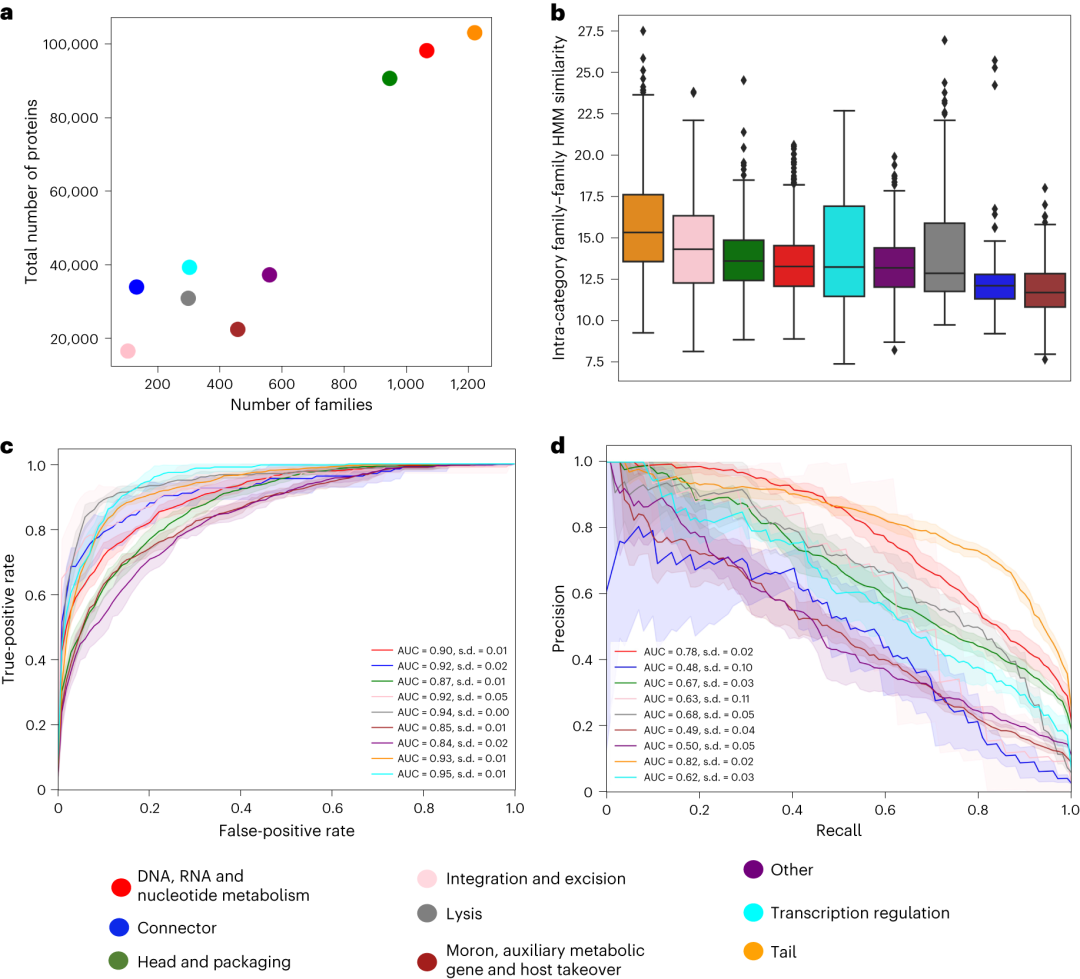
图2:使用基于 PLM 的蛋白质嵌入对 PHROG VPF 进行功能类别分类。a, PHROG类别家族和总蛋白数;
b, 功能类别中家族的成对谱相似性分布。方框代表四分位数范围;须线代表除异常值(菱形)之外的整个分布;水平线表示中位数。
(c-d)带注释的 PHROG 家族五重分层分割的多级函数分类器性能。
c, 平均AUC和s.d.超过5倍的接收器特性曲线。d, AUC和sd大于5倍的Precision-recall曲线。每折叠一次,对一个家族中的所有蛋白质进行训练,并对来自测试家族的随机单个序列进行测试。利用Transformer_BFD PLM嵌入蛋白质序列,分类器由三隐层密集神经网络和softmax激活的输出层组成。
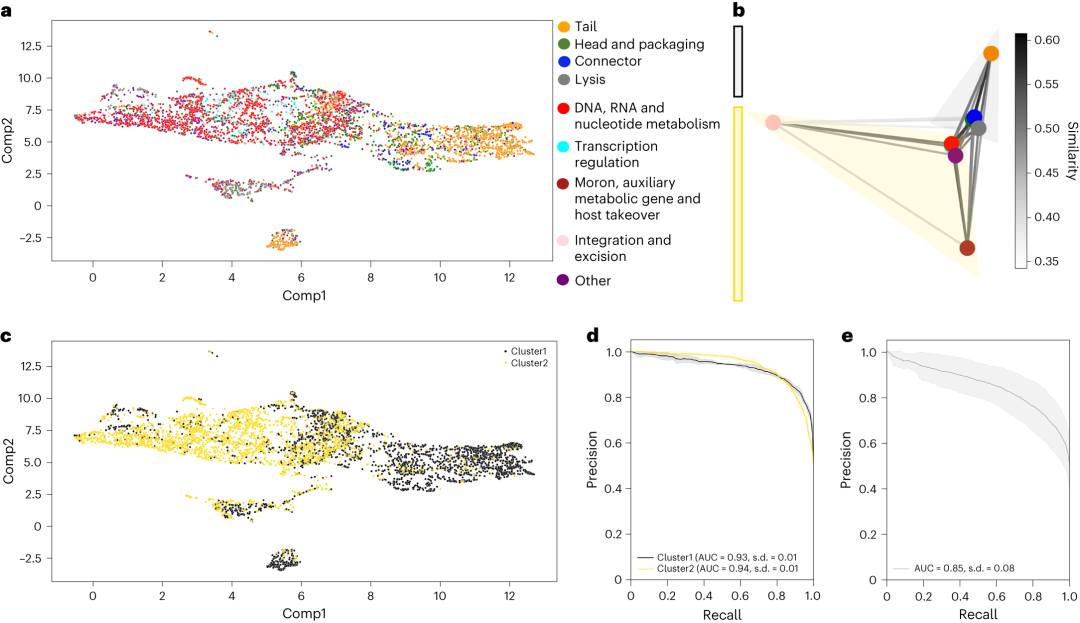
图3 基于 PLM 的 PHROG VPF 的嵌入研究。
a,PHROG VPF 的 UMAP 投影。VPF 表示为序列向量的质心。b,家族-家族间相似性(边缘权重)的光谱网络可视化,其测量为两个类别之间所有家族对的平均家族-家族质心相似性。类别-类别相似度矩阵以 n = 2 聚类为两组(黑色和黄色)。
c,光谱簇用于对 PHROG VPF UMAP 投影进行着色。
d,簇被用作PHROG VPF分类器的二进制类别,如图 2d 所示。
e,10 个随机 2 组分割的分类器性能,AUPRC 对组和分割进行平均。在 d 和 e 中,性能报告为五倍以上的平均 AUC,误差表示1 s.d,Comp1 和 Comp2 指 UMAP 投影维度。
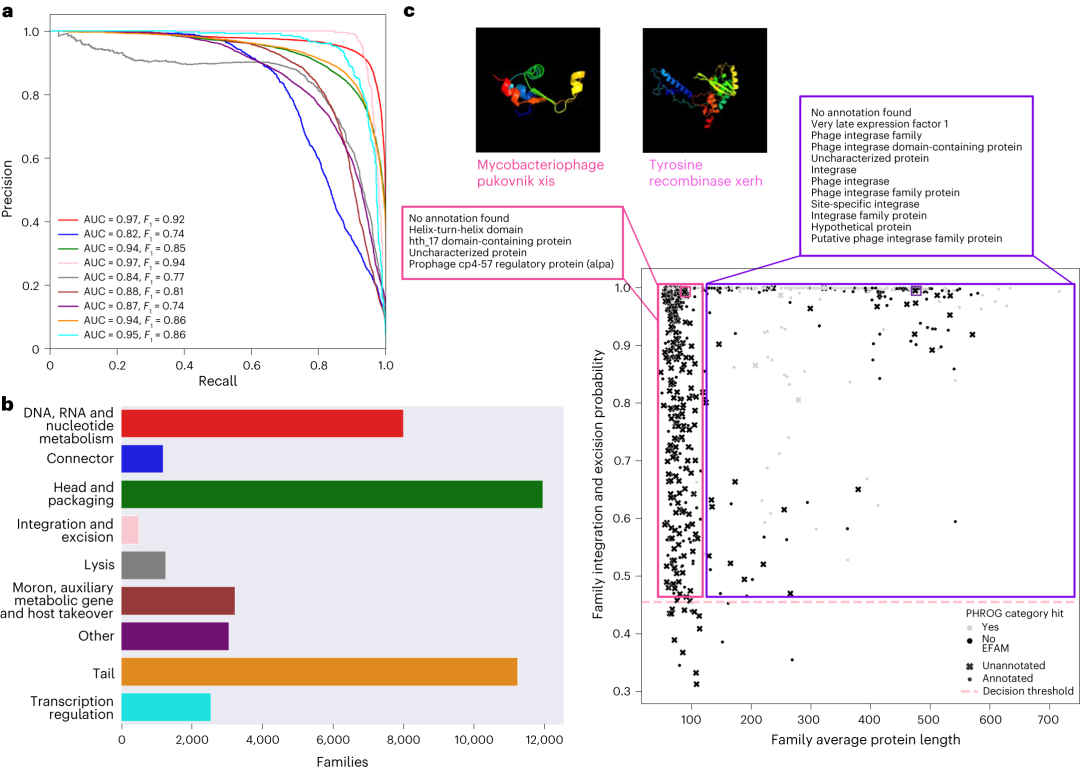
图4 使用从海洋病毒组中挑选的VPF的EFAM数据库进行功能类别分类器验证和发现。
a,用PHROGs HMM标记的Efam VPF的精度-回归曲线,并用基于PLM的函数分类器进行预测。表现是通过AUPRC和F1分数来衡量的。
b,EFAM中根据特定类别的最佳阈值标记到每个功能类别且未被PHROGs 的隐马尔柯夫模型捕获的VPF数量。
c,Efam VPF通过VPF中的平均蛋白质长度来预测整合和切除簇的概率。切除酶(粉红色)和整合酶(紫色)的注释,术语用于在EFAM(⋅)中注释的VPF。对于两个与PHROGs的隐马尔柯夫模型不匹配且未在Efam(×)中注释的Efam VPF的结构预测显示了预测的结构,一个切除酶(Cluster122519)和一个整合酶(Cluster86903)。决策概率是基于FDR的“整合和剔除”预测的门槛。
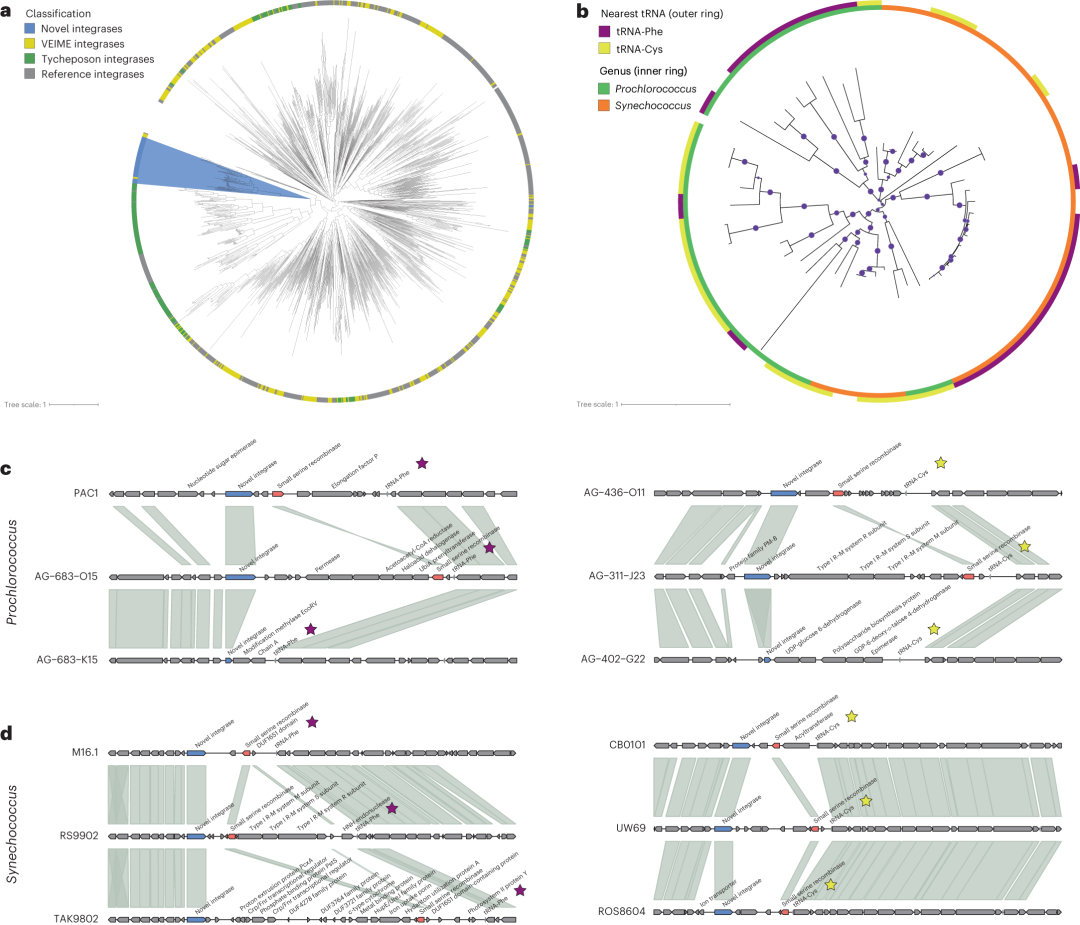
图5 海洋picocyanobacteria中酪氨酸整合酶的鉴定。
a,已鉴定的整合酶(蓝色)与海洋病毒寄生虫(VEIMEs;黄色)、蓝藻移动元件整合酶(绿色)中描述的酪氨酸重组酶的系统发育关系,以及在描述良好的噬菌体和移动元件中常见的类别(例如,噬菌体诱导的染色体岛,整合和共轭元件,以及整合子)。
b, 原绿球藻和聚藻球菌全长整合酶(>350个氨基酸)的系统发育分类,与最近的下游tRNA(外环)和基因组分类(内环)有关。由于基因组组装的限制,缺口反映了未知的tRNA关联。紫色点表示分支支撑度为0.5或更大。
c,d,海洋原绿球藻(c)和聚球藻(d)基因组中整合酶的基因组背景。有颜色的基因表示整合酶(蓝色),一个小丝氨酸重组酶经常在整合酶(红色)和下游tRNA(紫色或黄色星号)附近发现。阴影区域连接同源基因。
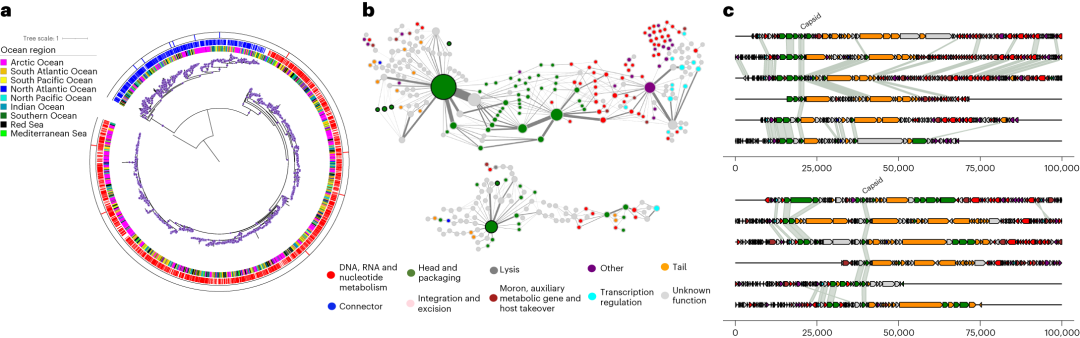
图6 一种主要衣壳蛋白(MCP)的发现。
a, MCP的系统发育和在全球海洋中的分布。MCP 有两个主要分支(红色和蓝色)。紫色圆点表示分支支撑度为 0.5 或更大。
b,包含重叠群的 MCP 的网络描述,其中蛋白质簇 (PC)(描述为节点)由包含 MCP 的所有重叠群构建,并计算两个 PC 在重叠群上相邻的次数(描述为边缘)。为了便于进行可视化,将针对大小 ≥10 个成员筛选PC。节点大小反映了 PC 中蛋白质的数量,边缘宽度反映了 PC 相邻的次数,节点上的黑色光晕表示 PC 是 MCP 的簇。
c, MCP在两个MCP分支中选定重叠群中的基因组背景。节点 (b) 和基因 (c) 的颜色表示基于 PLM 的功能分类器预测的功能类别。网络和基因组图谱代表顶部(红色)和底部(蓝色)的两个分支。
参考文献
Flamholz, Z.N., Biller, S.J. & Kelly, L. Large language models improve annotation of prokaryotic viral proteins. Nat Microbiol (2024). https://doi.org/10.1038/s41564-023-01584-8
- 通讯作者简介 -

阿尔伯特·爱因斯坦医学院
Libusha Kelly
副教授
Libusha Kelly 是阿尔伯特·爱因斯坦医学院系统与计算生物学和微生物免疫学部的副教授。Kelly在斯坦福大学获得人类生物学专业的学士学位,然后在加州大学旧金山分校Andrej Sali组攻读计算生物学方面的博士,并在麻省理工学院Sallie W. Chisholm组进行博士后研究。她的研究领域集中在微生物生态学,特别是微生物群落如何影响人类健康和全球海洋过程。Kelly教授在其实验室内进行了多项研究,包括探索不同个体的微生物组如何影响抗癌药物伊立替康的活化,发现海洋中广泛存在的新型细菌病毒(噬菌体)并研究了微生物在人体药物代谢中的作用。她还关注于阴道微生物多样性如何影响健康和疾病,以及如何有针对性地设计微生物群落以改善健康。
宏基因组推荐
猜你喜欢
iMeta高引文章 fastp 复杂热图 ggtree 绘图imageGP 网络iNAP
iMeta网页工具 代谢组MetOrigin 美吉云乳酸化预测DeepKla
iMeta综述 肠菌菌群 植物菌群 口腔菌群 蛋白质结构预测
10000+:菌群分析 宝宝与猫狗 梅毒狂想曲 提DNA发Nature
一文读懂:宏基因组 寄生虫益处 进化树 必备技能:提问 搜索 Endnote
16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流快速解决科研困难,我们建立了“宏基因组”讨论群,己有国内外6000+ 科研人员加入。请添加主编微信meta-genomics带你入群,务必备注“姓名-单位-研究方向-职称/年级”。高级职称请注明身份,另有海内外微生物PI群供大佬合作交流。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍未解决群内讨论,问题不私聊,帮助同行。
点击阅读原文,跳转最新文章目录阅读
更多推荐
 已为社区贡献17条内容
已为社区贡献17条内容







所有评论(0)